Colocation des données
Une problématique qui s’est posé est de pouvoir exécuter le code au plus près du stockage des données afin de limiter les transferts.
Étape 1 : répartition des données
Les fichiers CSV en entrée sont d’abord parsés puis divisés en plusieurs partitions sauvegardées sous forme de plusieurs fichiers sur HDFS (stockage distribué). Pour rappel de l’architecture actuelle : chaque machine exécute un datanode HDFS (stockage) et un worker Spark (exécuteurs). Le but est donc de répartir uniformément les partitions afin que chaque worker Spark ait un accès local au même nombre de partitions. De plus il faudrait que les mêmes numéros de partitions des différents catalogues soient stockées sur un même noeud (car les partitions sont ordonnées selon leur numéro healpix).
J’ai donc écrit un script en python permettant d’indexer les différentes partitions d’un fichier donné puis de les ordonnées de façon uniforme.
Fonctionnement :
- Accède à l’API HDFS et construit la liste des blocs du fichier donnés associés au noeud sur lequel il se trouvent
- Démare un serveur web en python sur chaque machine avec un accès au chemin de stockage des blocs
- Execute un find sur chaque noeud et construit un tableau associant les machines au blocs stockés (afin d’avoir le chemin non donné par l’API)
- Stoppe le service HDFS
- Pour chaque élément du tableau obtenu en 1) télécharge le bloc sur le bon noeud (avec curl) et supprime le fichier d’origine.
- Démare le service HDFS
Problèmes rencontrés
J’ai d’abord tenté de faire les transferts de données en utilisant netcat (c’est à dire démarer une instance en écoute et une deuxième en envoi). Cependant cette méthode résultait en de nombreux blocs corrompus, j’ai donc opté pour la solution plus simple de démarer un serveur web sur chaque noeud.
J’ai tenté de faire fonctionner ce script en suivant le patern producteur consommateur afin de le faire fonctionner en parallèle, cependant le script utilisant des connections SSH, le fait d’en démarer plusieurs en même temps coduisait rapidement à des instabilitées. Il doit toujours être possible de paralléliser ce script en utilisant un autre méchanisme que SSH.
Étape 2 : Exécution du code
On pourrait penser que le temps d’exécution aurait diminué, pourtant les performances obtenues en exécutant un cross match sur des fichiers non réordonnés puis sur les fichiers ordonnés sont similaire. En observant les logs on peut voir que la plupart des jobs sont exécutés sans préférence de localisation :
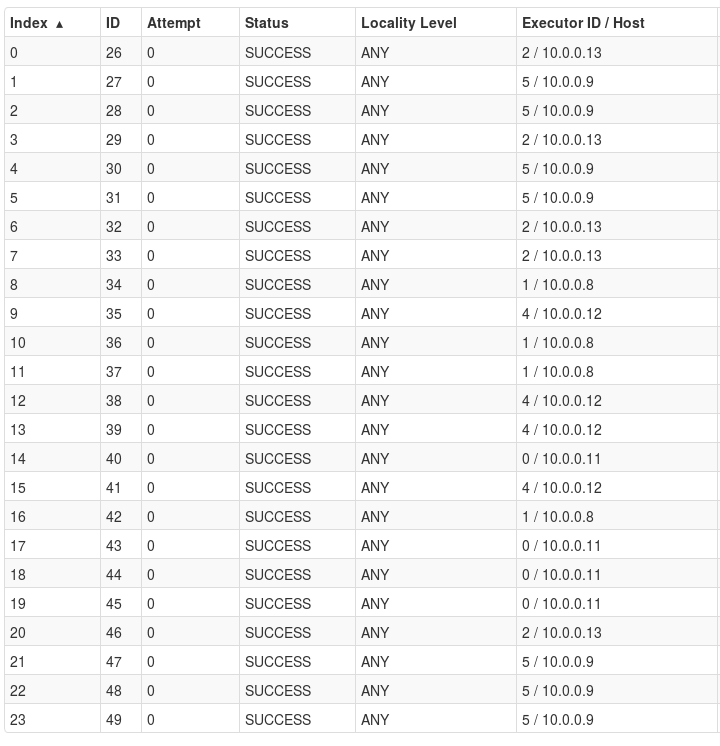
Résultats Spark standalone
Étape 3 : Utilisation de YARN
En recherchant des cas d’utilisations similaires sur internet il est souvent mentionné l’utilisation de YARN qui permettrait d’exécuter le code en fonction de la localisation des données.
Fonctionnement de YARN
Jusqu’à présent Spark fonctionnait en mode standalone, c’est à dire que tout fonctionnais uniquement en démarant des binaires Spark (Spark manager et Spark worker). En utilisant YARN on utiliserait des binaires Hadoop (YARN resource manager et YARN node manager). Les resource manager est le module servant à orchestrer le cluster et le node manager exécute les jobs reçu du resource manager. La différence par rapport à Spark est que lors de l’exécution d’un job spark les binaires Spark sont envoyés à chaque node manager. Le cluster Spark est donc créé à la volée et une fois le travail terminé tous les Spark workers sont supprimés. Il en résulte donc un temps supplémentaire au démarage pour transférer les binaires Spark et démarer les workers.
Résultats
Les logs de YARN nous indiquent que les taches sont donc bien réparties :
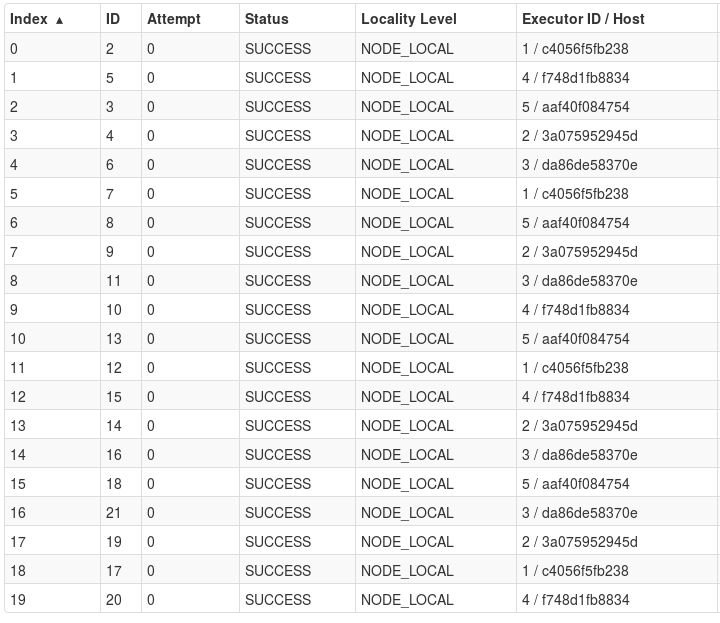
Résultats YARN
Cependant le temps d’execution reste le même et cela peu s’expliquer simplement : les différents noeuds utilisent des disques dur et le réseau est cablé en Gigabit, le débit du réseau est donc suppérieur au débit des disque dur, la vitesse de lecture est donc la même que l’on lise sur une machine distante ou sur un disque local. Si le réseau avait été en 100 Mbs ou que les machines fonctionnaient sur des SSD, la conclusion aurait sans doute été toute autre.
Perspectives
- Forcer le réseau à fonctionner en 100 Mbs (ou moins) pour confirmer que les performances sont améliorées dans le cas ou les disques sont plus rapides que le réseau
- Tester avec des SSD ou sur un service de cloud
Test de JupyterHub
Configuration
c.JupyterHub.port = 8765
c.JupyterHub.hub_ip = 'cds-stage-ms4'
c.Spawner.mem_limit = '2G'
c.JupyterHub.spawner_class = 'dockerspawner.DockerSpawner'
c.DockerSpawner.extra_create_kwargs = {'entrypoint': '/usr/local/bin/start-singleuser.sh'} # Ce script est necessaire dans le cas de jupyterhub
c.DockerSpawner.container_image = 'jupyter/minimal-notebook' # Image à utiliser
c.DockerSpawner.hub_ip_connect = 'cds-stage-ms4'Problèmes rencontrés
Le kernel python semble ne pas démarer correctement rendant impossible l’execution de code python